环境DNA分析的发展,为物种监测带来了新的可能性。

文|创瞰巴黎
导读
如何尽可能减少对野生动物生活的干扰,同时密切关注它们种群的变化。环境DNA(eDNA)分析的发展,有望解决这一难题。环境DNA分析,是一种不对目标物种造成危害的生物种群监测工具。对于加深人类对生物多样性的理解至关重要。
一览:
地球的生物多样性面临着重大危机 [1]。作为人类,我们既要密切关注野生动物种群的变化,又要尽可能减少对它们生活的干扰——二者看似矛盾,如何兼顾?所幸,随着环境DNA(eDNA)分析的发展,这一难题有望得到解决。
自上世纪八十年代起,科学家便开始利用新型测序技术和计算机技术,分析环境样本中的基因组,以辨别肉眼不可见的微生物。宏基因组学(直接研究样本中所有微生物基因组集合的学科)应运而生。
环境DNA不仅来自无处不在的微生物。所有生物不论大小,都会留下“蛛丝马迹”——黏液、死皮、毛发、尸骨、粪便等。这些物质所含的DNA信息可用于物种鉴定。2008年,法国国家科学研究中心高山生态学实验室验证了这一理论:学者们分析了多个水塘中的DNA物质,判断出了牛蛙活动的痕迹[2]。
这意味着环境DNA分析可作为监测大型生物种群的有效工具,在沉积物、海床、甚至空气中已得到成功应用。甚至有科学家发现,提取动物园的空气,分析其中的DNA物质,能推断出动物园内外的数十种物种,这一结果发表于2022年初的两篇学术论文中[3, 4]。环境DNA分析潜力无穷,但也存在一些缺陷。首先,让我们来了解其原理。
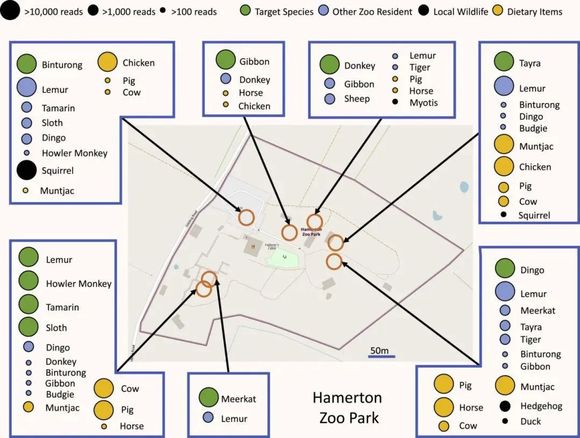
图片来源:新西兰汉密尔顿动物园空气eDNA分析结果
注:每种颜色对应一类物种(黄色为肉类饲料来源物种),圆圈大小代表检测结果推算出的种群规模。
分析环境样本中的DNA有三种方式。一是全局宏基因组测序:对样本中所有DNA物质进行测序,然后试图辨别已知物种的基因组。全局宏基因组测序适用于微生物研究,因为环境样本中的微生物DNA一般都相对完整,且含量较高。相比之下,大型生物的DNA在环境样本中含量低,支离破碎,片段长度只有几十至几千碱基对,不适合做全局宏基因组测序。
这时,就轮到条形码技术登场了。基因条形码,指可作为物种标记的基因片段。学者一般会选择较短的片段作为条形码,便于与环境中天然存在的破碎DNA匹配。样本里与已知条形码匹配的片段通过PCR扩增,便可进一步研究分析。条形码的定义可精准(指向一个物种)、可粗略(指向亲缘关系较近的多个物种);可监测具体物种,亦可统计一个区域的物种组成,前者称为条形码技术,后者称为宏条形码技术(一种具有针对性的宏基因组学手段)。
条码匹配鉴定,需使用已知物种的条形码数据库。不同细分领域的数据库有大有小,由此便引出环境DNA分析的一个局限性:有效的分析必须基于内容丰富的数据库。不过,随着学界收集的数据越来越多,这一障碍未来有望逐渐化解。
环境DNA分析现有四大应用:生物多样性研究(对一个区域的物种进行类别统计、长期监测、生理机能分析等[5])、物种跟踪检测(针对濒危物种、入侵物种、环境健康指示物种 [6])、种群规模估算、食性研究(通过分析粪便中的DNA [7])。
在海底沉积物中、极寒条件下,环境DNA可以保存成千上万年,蕴含古生物的奥秘。在阿拉斯加的布里斯托湾,研究者从环境中提取单细胞生物DNA,可追溯到1400年前,还能从其变迁看出二战和现代农业对环境的影响[8]。2022年12月初,研究者利用从格林兰永冻层采集的eDNA样本,重塑了200万年前的古生态系统,一举打破了记录[9]。
研究现代生物,环境DNA分析同样能派上大用场。环境DNA采集步骤十分简单,对环境不造成任何侵扰,无需复杂设备或培训,可用于难以直接观测种群的地区,可与现有科考项目轻易结合,且采集步骤与后续分析相对独立。低廉的成本、灵活的操作模式让大范围、长时间监测成为可能。大量样本统一分析,会形成“规模经济效应”,有利于分析工作的开展。
“在海底沉积物中、极寒条件下,环境DNA可以保存成千上万年,蕴含古生物的奥秘。”
但环境DNA分析也有其局限性。首先,在一个地方发现某物种的DNA,未必能证明该物种生活在该处。这一道理很简单,也很“致命”。再者,目标物种的健康状况、体积、发育阶段只能通过直接观测得知,无法通过环境DNA得出。DNA很容易在环境中迁移。河流生物的DNA,在其实际栖息地数公里开外仍能测出,所以环境DNA不能用来对种群精确定位。
不同物种在环境中留下的DNA量有多有少。若包裹DNA的细胞结构不够坚固,或者环境恶劣(pH、温度极高极低),DNA很容易快速分解。环境中检测不出DNA,不能说明不存在相应的物种。即使检出DNA,也有可能来自污染源:样本采集者、设备、环境都会污染样本。举个例子:有些沿海地区的餐厅、市场能检测出异地鱼类的DNA [10]。
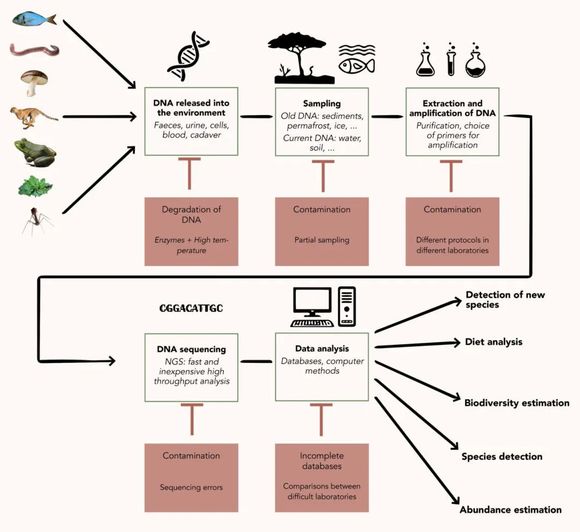
图片来源:Paul Castagné、Garance Castino所著文章 [11]
注:环境DNA分析流程图,绿框为具体步骤,红框为每个步骤的局限性。
检测的结果偏差,还来源于分子生物学研究手段和数字工具本身。一方面,现有的基因数据库尚不完整。另一方面,并非所有DNA片段都能有效地通过PCR扩增。若条形码选择不当或条形码辨别有误,则会出现假阳性、假阴性结果。项目样本量大的情况下,逐个验证结果有困难,何况采样过程、基因测序都会出现偏差,故环境DNA分析作为量化工具的适用性有限。每项环境DNA研究,都必须对结果进行仔细的核验,并与实地观察统计出的种群规模进行比对。
上述挑战是环境DNA学者的重点攻克对象。现已有数个颇具潜力的研究团队正在通过操作标准化、数据库扩充、条形码改良等方式降低环境DNA分析的不准确性[12]。
纽约城市大学博士生、鱼类种群环境DNA研究者Sam Chew Chin将环境DNA分析工具称作“基因鼻”——“嗅探”生态圈的新型手段。虽然不尽完美,但其解锁的种种可能性与其他工具相结合,能加深人类对生物多样性的了解。
参考资料
1.https://report.ipcc.ch/ar6wg2/pdf/IPCC_AR6_WGII_FactSheet_ Biodiversity.pdf
2.https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2610135/
3.https://www.sciencedirect.com/science/article/pii/S0960982221 016900
4.https://www.cell.com/current-biology/fulltext/S0960-9822(21)01650 X
5.Un exemple: https://www.ajspi.com/communiques-club/climat-le-role-mesestime-de-la-biodiversite-des-abysses-dans-la-pompe-a-carbone-oceanique/
6.Un exemple: https://www6.inrae.fr/synaqua/
7.Etude du régime alimentaire de l’apron du Rh ne https://hal.science/hal-02462400
8.https://www.cell.com/current-biology/fulltext/S0960-9822(21)00452–8
9.https://www.nature.com/articles/s41586-022–05453 y
10.https://youtu.be/WHcY9d6oq4A?t=2630
11.https://planet-vie.ens.fr/thematiques/ecologie/ l adn-environnemental-un-nouvel-outil-pour-espionner-les-especes-sauvages
12.Exemples d’un projet européen http://dnaqua.net/ about/ et d’un projet canadienhttps://itrackdna.ca/ index.php/buts-objectifs/?lang=f